所內動態
Data Is All You Need: 提升AI模型泛化能力的BindingNet v2
在圖像識別領域,ImageNet數據集的重要性不言而喻。在生物計算領域,2024年黃牛實驗室發布的BindingNet v1數據集獨樹一幟,采用模板匹配技術構建了高質量的蛋白-配體復合物三維結構模型,有效減輕了AI模型在蛋白-小分子親和力預測中因包埋溶劑可及表面積引發的偏見等問題(https://mp.weixin.qq.com/s/2KkD73ZC2pJh2-t24bpsWw),為蛋白-配體相互作用研究領域中AI模型的評估和優化奠定了堅實基礎,此階段我們稱之為“From Big Data to Good Data”。
然而,AI對數據的需求永無止境,接下來我們進入 “Make Good Data Greater”的階段。2025年1月8日,北京生命科學研究所/清華大學生物醫學交叉研究院黃牛實驗室于《npj Drug Discovery》雜志在線發表題為“Augmented BindingNet dataset for enhanced ligand binding pose predictions using deep learning ”的研究論文。在BindingNet v1數據集的模板匹配技術基礎上,創新性地提出了基于片段化結合形狀與靜電匹配的多層次模板匹配流程,成功構建出規模遠超BindingNet v1近十倍規模的蛋白-配體相互作用數據集— BindingNet v2。此數據集覆蓋1794個蛋白靶點,包含689,796個蛋白-小分子配體復合體結構模型及其相應的實驗活性數據,極大地提升了AI模型在蛋白-配體復合體預測任務上的泛化能力。尤其在僅使用Tc < 0.3的數據(不包含同測試集中結構類似的分子)訓練時,Uni-Mol模型在PoseBusters測試集上的泛化能力從38.55%大幅躍升至74.07%。
01
多層次的模板匹配流程
研究團隊從 PDB 數據庫中篩選出 26,438 套高質量的蛋白-小分子復合物結構作為模板,并從 ChEMBL 數據庫中挑選出 724,319 對實驗驗證的蛋白-小分子數據對。隨后,通過以下五個步驟構建多層次模板匹配流程(圖1):
1.計算候選分子與模板分子之間的最大公共子結構占有率。
2.關鍵子結構疊合:
a)若候選分子的最大公共子結構占有率超過 0.6,則直接將候選分子與模板分子進行疊合;
b)若未達到占有率要求,則對候選分子進行片段化處理,并借助SHAFTS 工具探尋片段與模板分子間三維形狀及藥效團疊合程度(即 hybrid score)最高的構象。
3.構象采樣:使用ETKDG對疊合后的剩余部分結構進行采樣、聚類、過濾操作,計算hybrid score,以確保構象的合理性。
4.能量最小化:挑選hybrid score排名前20個的復合物結構進行MM/GB-SA優化。
5.打分:選取 hybrid score 最高的復合體作為最終構象。
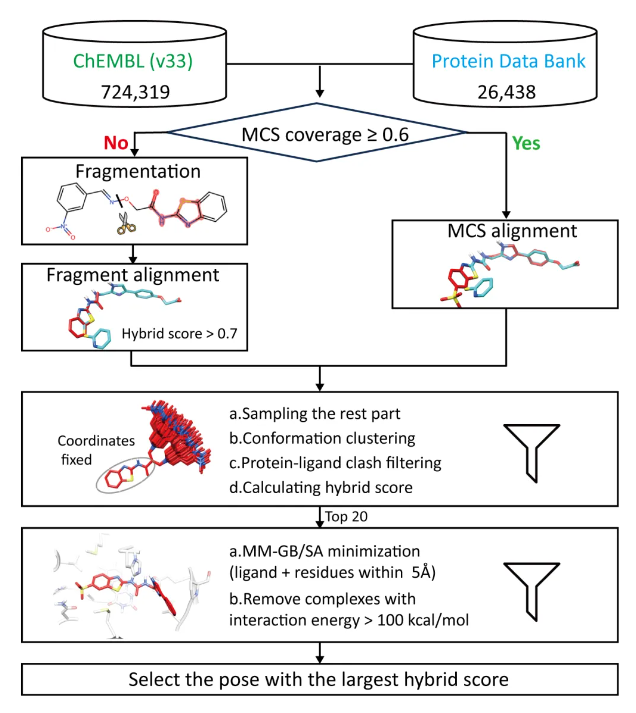
圖1:多層次模板匹配流程
02
BindingNet v2數據集與網站介紹
借助多層次的模板匹配流程,研究團隊成功構建了689,796個蛋白-小分子復合物結構,并為每個復合物附上對應的實驗活性數據。同時,依據 hybrid score 對數據集進行質量分級,其中高質量(hybrid score ≥ 1.2)、中質量(1.0 ≤ hybrid score < 1.2)、低質量(hybrid score < 1.0)的數據分別占33.63%、23.91%、42.45%。為便于科研人員檢索和分析,團隊搭建了專屬網站(http://bindingnetv2.huanglab.org.cn/),用戶可通過該網站查看構建的復合物三維結構,開展蛋白-小分子相互作用分析。
03
BindingNet v2對深度學習模型泛化能力的顯著提升
盡管已有多個深度學習模型在 PoseBusters 測試集上展現較高的成功率(如 Uni-Mol v1:62.4%、AlphaFold3 with pocket specified:90.0%),但本研究團隊發現,這些模型所采用的訓練集與測試集之間小分子結構存在很高的相似性。例如,測試集中70.09%的樣本能在PDB(v2019)中找到相似性大于0.7的訓練樣本,超過90%的測試集樣本都能從PDB(v2019)中找到相似性大于0.3的訓練樣本。然而,在實際應用中,基于靶標結構的虛擬篩選最有意義的應用場景是發現全新骨架的小分子,且通常以 Tc < 0.3 作為最嚴格的篩選標準。因此,研究團隊僅使用Tc < 0.3的訓練樣本嚴格評估Uni-Mol的泛化能力。結果顯示,僅用 PDBbind(Tc < 0.3) 數據訓練時,Uni-Mol 模型在 PoseBusters 測試集上的成功率僅為 38.55%;隨著BindingNet v2 中Tc < 0.3 的數據逐步加入訓練集,模型泛化能力顯著提升至 64.25%;結合MM/GB-SA優化和重打分后,成功率進一步提升至 74.07%,并通過了所有物理化學合理性檢查(圖2)。值得關注的是,僅用 Tc < 0.3 數據訓練的模型在類藥子集上的成功率已超越用PDB(v2019)訓練的AlphaFold3模型,充分驗證了BindingNet v2 數據集的重要價值。然而,BindingNet v2中的蛋白和小分子結構多樣性仍然受限于PDB數據庫,未來,黃牛實驗室將通過整合多層次數據、深度學習預測復合體結構、結合物理方法優化結構、以及引入半自動化質量標注流程等多方面的迭代優化,為蛋白-配體相互作用研究領域提供更全面且高質量的數據支持,進而提升人工智能方法在柔性對接、復合體構象動態過程預測以及蛋白-小分結合能預測等方面的表現。
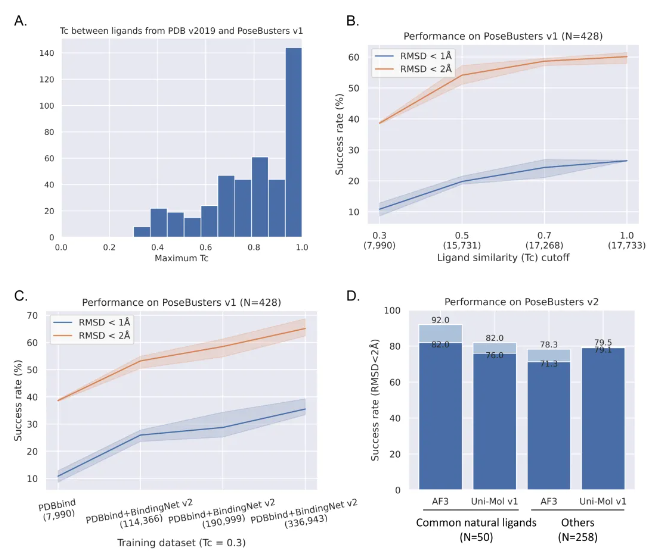
圖2:使用BindingNet v2訓練Uni-Mol
黃牛實驗室的博士研究生朱慧為本文第一作者,黃牛博士為通訊作者。其他作者包括黃牛實驗室的李雪蓮博士和陳保全。ByteDance AML - AI for Science Team為本研究提供了重要的算力支持。該項研究獲得北京市科委和清華大學資助,在北京生命科學研究所完成。此外,特別感謝李偉博士對本文相關內容撰寫方面給予的重要幫助。
論文鏈接
https://doi.org/10.1038/s44386-024-00003-0
BindingNet v2網站:http://bindingnetv2.huanglab.org.cn/
BindingNet v1:https://pubs.acs.org/doi/10.1021/acs.jcim.3c01170



